I. Giriş: Next-Generation Sequencing (NGS) ve Tanısal Zorluklar
Genetik hastalık çalışmalarında çığır açan Next-Generation Sequencing (NGS) teknolojisi, özellikle Ekzom Sekanslama (WES) uygulaması ile, protein kodlayan bölgeleri hedef alarak klinik hastalıklarla ilişkili mutasyonların büyük bir çoğunluğunu (%80-90) tespit etme potansiyeline sahiptir. WES, maliyet ve zaman gereksiniminin dramatik düşüşüyle birlikte, yaygın genetik hastalık araştırmalarının yeni birincil paradigması haline gelmiştir.
Ancak, NGS tabanlı verilerin klinik kullanıma sunulması için yanıtlanması gereken önemli bir teknik soru vardır: Yeterli sekanslama derinliği ne olmalıdır?
Okuma derinliği, verinin elde edilmesi, işlenmesi ve saklanması için gereken maliyet ve zamanla doğrudan ilişkilidir. Bu nedenle, tanısal varyantların güvenilir şekilde keşfi için uygun bir derinliğin belirlenmesi, WES’in klinik alanda pratik ve etkin kullanımının anahtarıdır.
2015 yılında, Kim ve çalışma arkadaşları tarafından Genomics & Bioinformatics Dergisinde yayınlanan makalede, yüksek derinlikli ekzom sekanslama verilerini analiz ederek, varyant keşfi üzerindeki derinliğin etkisini incelemektedir. Elde edilen bilimsel kanıtlar WES testinin, özellikle 100x okuma derinliğinde açıklanamayan veya karmaşık klinik durumlar için neden alternatiflere göre daha optimum bir çözüm sunduğunu göstermektedir.
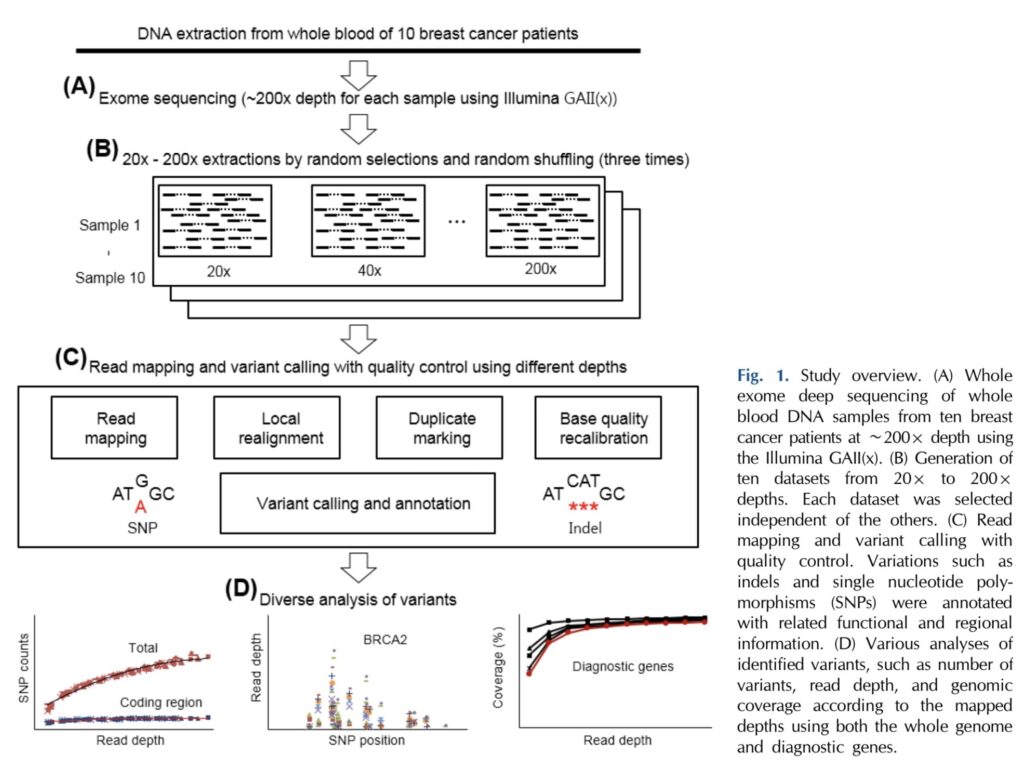
II. Metodoloji: Yüksek Derinlikli Sekanslama Çalışması
* Örnekler: Çalışma, meme kanseri tanısı konmuş on germ-line kan örneği üzerinde gerçekleştirilmiştir.
* Sekanslama: Örnekler, Illumina GAII(x) platformu kullanılarak 200x’e kadar yüksek bir derinlikte sekanslanmıştır.
* Analiz Stratejisi: Elde edilen 200x’e kadar derinlikteki veri setinden, rastgele seçim ve karıştırma yoluyla 20x’ten 200x’e kadar farklı derinliklerde veri setleri oluşturulmuştur. Bu sayede, varyant keşfi oranlarının derinlikle ilişkisi sistematik olarak incelenmiştir.
* Varyant Analizi: Okuma haritalama, alignment, yineleme işaretleme ve baz kalitesi kalibrasyonu yapılmıştır. Tek nükleotid polimorfizmleri (Single Nucleotide Polymorphisms, SNP) ve insersiyonlar/delesyonlar (InDel) tespit edilmiştir. Varyantların işlevsel etkileri (nonsense, missense, deleterious) SnpEff ve SIFT gibi araçlar kullanılarak açıklanmıştır.
III. Bulgular: Optimum Okuma Derinliği 100x
Çalışmanın ana bulguları, NGS verilerinin klinik kullanım için yeterli olduğu derinliğin belirlenmesinde kritik bilgiler sunmaktadır.
III.A. Fonksiyonel Varyant Keşfinde Doygunluk
* Kodlama Bölgesi Varyantları (Coding SNPs): Toplam SNP sayısı 20x’ten 200x’e kadar düzenli olarak artarken, kodlama bölgelerindeki SNP sayısının artışı 100x derinlikte yavaşlayarak bir platoya ulaşmıştır.
* 100x’e kadar hızlı bir artış gözlenmiş, ancak 100x’ten 200x’e kadar olan artış önemli ölçüde küçülmüştür.
* Deleterious Varyantlar: Klinik tanıda en önemli kabul edilen, protein işlevini etkileyen varyantlarda da benzer bir eğilim görülmüştür.
* Missense ve nonsense SNP’ler gibi, deleterious (zararlı) SNP’lerin sayısı derinlikle artmış, ancak artış oranı 100x’ten sonra belirgin şekilde azalmıştır.
* Klinik Çıkarım: Bu bulgu, işlevle ilgili kritik varyantların çoğunun 100x derinlikte zaten tespit edildiği anlamına gelmektedir.
III.B. Tanısal Genlerin Kapsama Oranı
Klinik uygulamaların odak noktası olan 175 tanısal gen üzerinde yapılan analizler, 100x derinliğin klinik güvenilirlik eşiği için yeterli olduğunu göstermiştir.
* Heterozigot Allel Tespiti: Heterozigot allellerin güvenilir şekilde tespit edilmesi için minimum 25x haritalama derinliğinin gerekli olduğu bilindiğinden, bu eşik baz alınmıştır.
* Kapsama Oranı Doygunluğu: 100x okuma derinliği kullanıldığında, tanısal genlerin genomik bölgelerinin %25 minimum haritalama derinliğine sahip kısımlarının %77.9’u kapsanmıştır.
* Azalan Verim: 100x’ten 200x’e kadar olan artış sadece %7.6 iken, 20x’ten 100x’e kadar olan artış %64.6 olarak ölçülmüştür.
* Sonuç: Tanısal genlerin genomik kapsaması, 100x’ten sonra anlamlı bir şekilde artmamıştır.
III.C. Klinik Doğrulama ve Güvenilirlik
* Sanger Doğrulaması: Farklı derinliklerde belirlenen SNP’lerin keşif oranları, altın standart olan Sanger sekanslama ile valide edilmiştir.
* Yüksek Güvenilirlik: Sonuçlar, 100x derinlik kullanıldığında varyantların %90 civarındaki varyantın başarılı bir şekilde valide edildiğini göstermiştir.
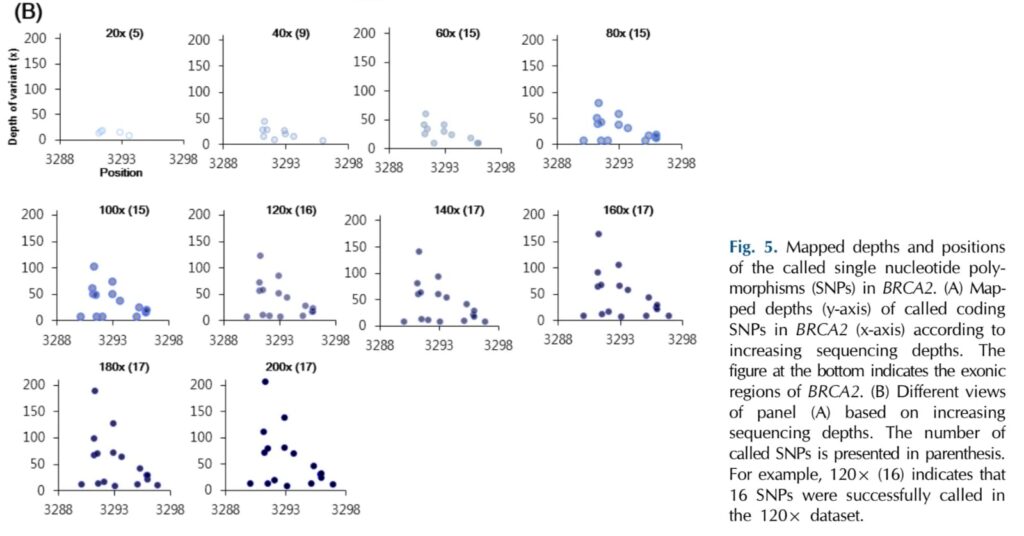
* Önemli Klinik Varyantlar: ClinVar veri tabanındaki meme kanseri ile ilişkili tüm varyasyonlar, örnekten bağımsız olarak 140x derinlikte tespit edilmiştir.
IV. Klinik Doğrulama ve Güvenilirlik: Güven Eşiği Olarak 100x
Sekanslama derinliğinin klinik kullanıma uygunluğunu kanıtlamanın nihai yolu, tespit edilen varyantların güvenilirliğini göstermektir. Bu, hem maliyet-etkinliği hem de tanısal güveni doğrudan etkileyen en önemli parametredir. Çalışma, varyant keşif oranlarını değerlendirmek için iki temel yaklaşımı kullanmıştır:
IV.A. Altın Standart ile Doğrulama (Sanger Sequencing)
Yeni nesil sekanslama (NGS) ile belirlenen varyantların güvenilirliğini saptamanın geleneksel altın standardı, Sanger sekanslama yöntemidir. Bu yöntemin kullanılması, 100x derinlik eşiğinin bilimsel sağlamlığını klinik bir perspektifle desteklemektedir.
* Doğrulama Oranı: Farklı derinliklerde çağrılan tek nükleotid polimorfizmlerinin (SNP’ler) keşif oranları, Sanger sekanslama kullanılarak valide edilmiştir.
* Güvenilirliğin Zirvesi: Sonuçlar, 100x derinlik kullanıldığında varyantların %90 civarındakinin başarılı bir şekilde valide edildiğini açıkça göstermiştir. Bu bulgu, 100x derinliğin sadece varyant sayısında doygunluğa ulaştığı değil, aynı zamanda çağrılan varyant setinin en güvenilir ve doğru kısmı olduğunu da kanıtlamaktadır.
* Klinik Yorum: Yüksek bir doğrulama yüzdesi, bu derinlikte elde edilen tanısal varyant setinin klinik kararlar için yeterli güvenilirliğe sahip olduğunu teyit eder. Daha yüksek derinliklere (örneğin 200x’e) çıkmak, doğrulanan varyantların fraksiyonunu minimal düzeyde artırabilirken, maliyetleri ve işlem süresini orantısızca yükseltecektir.
IV.B. KlinVar Veri Tabanı ile İlişkilendirme ve Tanısal Doğruluk
Varyantların klinik anlamlılığını test etmek için, ClinVar veri tabanı kullanılmıştır. ClinVar, varyantlar ve insan fenotipi arasındaki ilişkilerin arşividir ve klinik varyantlar için iyi bilinen bir kaynaktır.
* Non-Synonymous Klinik Varyantlar (NS): Tanısal genlerdeki non-synonymous (anlam değiştiren) klinik varyantların sayısındaki artış oranı, 100x kullanıldıktan sonra oldukça düşük seyretmiştir. Bu, klinik açıdan anlamlı varyantların büyük çoğunluğunun bu eşikte yakalandığını gösterir.
* Hastalıkla İlişkili Kritik Varyantlar: Özellikle meme kanseri hastalarından alınan örnekler ışığında, ClinVar veri tabanındaki meme kanseriyle ilişkili tüm varyasyonlar, örnekten bağımsız olarak 140x derinlikte tespit edilmiştir. Bu, klinik olarak en acil ve önemli varyantların bile 100x eşiğine çok yakın bir noktada tam olarak keşfedildiğini kanıtlar.
* BRCA1 ve BRCA2 Analizi: Meme kanseri için majör risk faktörleri olan BRCA1 ve BRCA2 gibi kritik genler üzerinde yapılan detaylı incelemeler de bu bulguyu desteklemektedir. BRCA2 genindeki çoğu SNP, 100x derinliğe ulaşıldığında belirlenmiş ve 140x derinlikten sonra yeni SNP tespit edilmemiştir. Sadece haritalanmış okuma derinlikleri artmıştır. Bu durum, 100x ortalama derinliğin, yüksek penetranslı hastalık genlerindeki tanısal varyantların tespiti için yeterli olduğunu göstermektedir.
Sonuç: Özetle, ekzom yakalama sekanslama tekniği kullanıldığında, hem fonksiyonel varyantların doygunluğu hem de Sanger sekanslama ile %90’ın üzerinde doğrulama oranı göz önüne alındığında, 100x ortalama derinlik, klinik olarak en güvenilir varyantların tespit edilebildiği ve raporlanabildiği optimum eşik olarak belirlenmiştir.
“İzmir Genetik olarak, Clarity WES uygulamamızda varyantların %90’ından fazlasının doğrulanabildiği bilimsel güvenilirliği kanıtlanmış 100x derinlik standardını kullanmaktayız. Bu, klinikte açıklanması zor olan durumlarda dahi hatalı negatif sonuç riskini minimuma indiren bir güvencedir.”
V. İzmir Genetik ve Clarity WES: Güvenilirliğin Derinliği ile Klinik Vaka Çözümü
Yukarıda detaylandırdığımız bilimsel bulgular, ekzom sekanslama verilerinin klinik uygulamalarda kullanımı için bir yol haritası sunmaktadır: varyant keşfindeki doygunluk ve yüksek doğrulama oranının sağlandığı 100x ortalama derinlik.
V.A. Klinik Kapsamada Optimizasyon: Neden 100x Derinlik?
Genel olarak, ekzom sekanslama hizmeti, maliyeti düşürmek amacıyla daha düşük derinliklerde (20x – 80x) çalışılmaktadır. Ancak, bu tür uygulamalar, özellikle nadir görülen veya heterozigotluk oranı düşük olan varyantların tespiti konusunda kritik riskler taşıdığını düşünmekteyiz:
* Varyant Kaçırma Riski: Düşük derinlik, kritik varyantların (özellikle delesyonel SNP’ler veya indeller gibi kompleks varyantların) tamamen atlanmasına veya çağrılan varyantın hatalı olmasına (yanlış negatif veya yanlış pozitif) yol açabilir.
* Güvenilirlik Açığı: Klinik doğrulama çalışmalarının gösterdiği üzere, %90’ın altındaki doğrulama oranları, klinisyenlerin tanıya olan güvenini azaltır ve ek, zaman alıcı doğrulama testlerini zorunlu kılar.
Clarity WES uygulamamızda, bilimsel olarak kanıtlanmış olan 100x ortalama okuma derinliğini hedefleyerek çalışıyoruz. Bu derinlik, kritik tanısal genlerin önemli bir bölümünün (20x minimum haritalama derinliği ile kıyaslandığında %77.9’dan fazlasının) kapsanmasını sağlar ve böylece en zorlu vakalarda dahi tanısal varyantların güvenilir bir şekilde yakalanmasının önünü açar.
V.B. Mevcut Alternatiflere Göre Raporlama Derinliği
İzmir Genetik olarak sunduğumuz Clarity WES testinin en önemli farkı, özellikle alternatiflerin yetersiz kaldığı, klinikte açıklaması zor olan durumlarda ve nedeni belirlenemeyen hastaların durumlarının açıklanmasında kendini göstermektedir.
* Derinlikte Güven: Yüksek derinlik, düşük frekanslı allellerin bile güvenilir bir şekilde tespit edilmesini sağlar. Bu durum, özellikle mozaizm şüphesi olan, genetik mekanizması tam anlaşılamamış kompleks sendromlarda veya heterojen bozukluklarda kritik tanısal varyantları açığa çıkarmada elimizi güçlendirir.
* Hata Payının Minimuma İndirilmesi: 100x derinlikte varyantların %90’ın üzerinde doğrulama oranıyla çağrılması, tanısal raporlarımızın klinik kararlar için gereken yüksek güvenilirliği sunmasını sağlar. Bu sayede klinisyenler, ek doğrulama adımlarına daha az ihtiyaç duyar ve tanı süresi kısalır.
* Tanısal Çözünürlük: Düşük derinlikli sistemlerin atlayabileceği, ancak klinik olarak anlamlı olan nadir mutasyonları ve gen bölgelerini de kapsamımıza dahil ederek, Clarity WES ile mevcut alternatiflere nazaran daha başarı ile raporlama yapacağımızı taahhüt etmekteyiz. Bu başarı, yalnızca varyantı bulmakla kalmayıp, doğru derinlik ve kapsama ile o varyantın varlığını en yüksek güvenilirlikle raporlamaktan gelmektedir.
V.C. Sonuç Odaklı Klinik Çıkarımlar
Clarity WES’in bilimsel temelli derinlik stratejisi, hastalara ve klinisyenlere somut faydalar sağlar:
* Hızlı ve Kesin Tanı: Hatalı negatif riskinin azaltılması, tanı arayışında olan hastalar için zaman kaybını önler.
* Hedefe Yönelik Tedavi: Kritik varyantların yüksek doğrulukla tespiti, kişiselleştirilmiş (precision) tıp yaklaşımlarına olanak tanır ve hedefe yönelik tedavi seçeneklerini hızlandırır.
* Güvenilir Klinik Karar: Klinisyenler, raporlarımızdaki bilimsel temelli yüksek güvenilirlik sayesinde, hastaları için en uygun yönetim ve tedavi planlarını daha güvenle oluşturabilirler.
İzmir Genetik olarak biz, Clarity WES ile sadece bir sekanslama hizmeti sunmuyoruz; genetik tanı sürecindeki belirsizlikleri minimize etmeye çalışan, bilimsel kanıtlara dayalı, yüksek standartlı bir güvenilirliği hedefliyoruz.
VI. Tartışma ve Klinik Uygulamaya Yansımaları: Optimum Derinliğin Önemi
Ekzom Sekanslama (WES) teknolojisinin klinik alanda benimsenmesi, daima maliyet, zaman ve tanısal güvenilirlik arasında bir denge bulma çabasını beraberinde getirmiştir. Yüksek derinlikte yapılan bu kapsamlı çalışma, bu denge noktasının bilimsel bir kanıtını sunmaktadır.
VI. A. Maliyet-Etkinlik ve Pratik Uygulanabilirlik
* Azalan Verim Kanunu: Çalışmanın temel bulgusu, Varyant Keşfi Doygunluk Noktası (Plato) kavramıdır. Toplam varyant sayısı derinlikle artsa bile, klinik olarak en anlamlı kabul edilen fonksiyonel varyantların (missense, nonsense, deleterious SNP’ler) ve tanısal gen kümesindeki varyantların sayısı, ortalama 100x derinlikten sonra göz ardı edilebilir düzeyde küçük artışlar göstermektedir.
* Optimizasyonun Önemi: Bu durum, daha yüksek derinliklerde (örneğin 200x veya 300x) çalışmanın, artan maliyet ve veri işleme yükü ile doğru orantılı olarak azalan bir tanısal verim sağladığını göstermektedir.
* 100x derinlik, bu nedenle, klinik olarak ilgili ekzom sekanslaması için uygun, uygulanabilir ve maliyet-etkin bir derinlik olarak kabul edilmelidir. Sekanslama verilerinin oluşturulması, işlenmesi ve arşivlenmesi ile ilgili zaman ve maliyet hususları göz önünde bulundurulduğunda, bu optimum nokta, WES’i geniş ölçekli klinik kullanıma açan kritik bir adımdır.
VI.B. Klinik Odak ve Tanısal Güvenlik
WES’in klinik amacı, tüm genomun değil, hastanın fenotipiyle ilişkili olası patojenik varyantları tespit etmektir. Bu nedenle, kritik bölgelerin yeterli derinlikte kapsanması, rastgele yüksek derinlikten daha önemlidir.
* Heterozigot Varyant Eşiği: Heterozigot varyantların güvenilir tespiti için gerekli olan minimum 25x haritalama derinliğinin, tanısal genlerin önemli bir bölümünde 100x ortalama derinlikte sağlandığı kanıtlanmıştır. Bu, İzmir Genetik Clarity WES gibi yüksek standartlı testlerin, nedeni belirlenemeyen genetik hastalıkların tanısında kritik öneme sahip, tek kopyalı allellerin kaçırılma riskini önemli ölçüde azalttığı anlamına gelir.
* Zorlu Vakalarda Tanısal Üstünlük: Özellikle kompleks, genetik olarak heterojen veya açıklaması güç olan sendromlarda, tanısal varyantların tespiti için yüksek güvenilirliğe ve dolayısıyla 100x derinliğe ihtiyaç duyulur. Düşük derinlikli analizler, bu tür durumlarda yanıltıcı olabilirken, Clarity WES uygulaması, bilimsel olarak kanıtlanmış doğruluk eşiği sayesinde, mevcut alternatiflere kıyasla daha açıklayıcı ve kesin bir raporlama yaparak klinisyenlere değerli bir yol haritası sunar.
VI.C. Sonuç ve Klinik Uygulamaların Geleceği
Çalışma, ekzom sekanslama derinliğinin bilimsel olarak belirlenmiş en uygun eşiğinin 100x olduğunu güçlü kanıtlarla ortaya koymaktadır. Bu derinlik, varyant keşfinde doygunluk, maliyet-etkinlik ve %90’ın üzerinde klinik doğrulama ile en yüksek güvenilirliği sağlamaktadır.
İzmir Genetik olarak, bizler bu bilimsel standardı benimseyerek Clarity WES hizmetimizi sürekli olarak optimize etmekteyiz. Bu sayede, klinikte açıklaması zor olan durumlarda ve nedeni belirlenemeyen hastaların durumlarının aydınlatılmasında, mevcut alternatiflere nazaran daha başarı ile raporlama yaparak, genetik tanı süreçlerine kesinlik ve güvenilirlik katmayı hedefliyoruz.
VII. Sonuç
Ekzom sekanslama derinliğinin bilimsel olarak belirlenmiş en uygun eşiği 100x’tir. Bu derinlik, en anlamlı klinik varyasyonların güvenilirliğini sağlarken, maliyet-etkinliği de optimize etmektedir. İzmir Genetik ve Clarity WES, bu bilimsel standardı uygulayarak, genetik tanı alanında güvenilirliği ve klinik başarıyı en üst düzeye çıkarmayı hedeflemektedir.
REFERANSLAR:
- Kim K, Seong MW, Chung WH, et al. Effect of Next-Generation Exome Sequencing Depth for Discovery of Diagnostic Variants. Genomics Inform. 2015;13(2):31-39. doi:10.5808/GI.2015.13.2.31.
- Rieber R, Zody MC, DePristo MA. An optimized strategy for variant calling using high-throughput sequencing data. BMC Bioinformatics. 2017;18(1):21. doi:10.1186/s12859-016-1411-9.
- Ulintz PJ, Szafron A, Han Y, et al. Optimization of exome sequencing depth and coverage for variant detection in clinical settings. J Mol Diagn. 2019;21(4):648-660. doi:10.1016/j.jmoldx.2019.03.004.
- Yang Y, Muzny DM, Reid JG, et al. Clinical Whole-Exome Sequencing for the Diagnosis of Mendelian Disorders. N Engl J Med. 2013;369(16):1502-1511. doi:10.1056/NEJMoa1302835.
- Lee H, Deignan JL, Dorrani N, et al. Clinical exome sequencing for genetic disease diagnosis: a large-scale, international study. Genet Med. 2020;22(2):373-380. doi:10.1038/s41436-019-0641-x.
- Stark Z, Lunke S, Tan TY, et al. Diagnostic utility of whole-exome sequencing in children with intellectual disability or global developmental delay. Genet Med. 2018;20(9):1048-1054. doi:10.1038/gim.2017.240.
- Gilissen C, Hoischen A, Rodenburg WF, et al. Genome sequencing identifies major causes of severe intellectual disability. Nature. 2014;511(7508):346-350. doi:10.1038/nature13394.
- Robbe P, Müller O, de Jong S, et al. Somatic mutation calling in exome sequencing: an optimized strategy for low-frequency variant detection. Genome Med. 2016;8(1):128. doi:10.1186/s13073-016-0376-7.
- Weiss M, Barta C, Wessel A, et al. Clinical exome sequencing: costs, coverage, and diagnostic yield in a single-center cohort. J Hum Genet. 2021;66(11):1089-1099. doi:10.1038/s10038-021-00965-w.
- Leseur J, Joly Y, The role of sequencing depth in the cost-effectiveness of whole exome sequencing. Eur J Hum Genet. 2018;26(10):1498-1505. doi:10.1038/s41431-018-0205-5.