Kanser tedavisinin geleceği, tek bir biyolojik katmanın analiz edilmesinden, hastalığın tüm moleküler profiline bütüncül bir yaklaşımla (Multi-Omic) haritalandırmaya doğru ilerlemektedir. Geleneksel olarak, tedavi kararları histopatoloji ve sınırlı genetik belirteçlere dayanıyordu. Ancak son dönemde yayımlanan çalışmalar, kanser hücrelerinin karmaşık doğasını anlamak ve kalıcı yanıtlar elde etmek için genomik, transkriptomik ve özellikle proteomik yaklaşımların entegre edilmesi gerektiğini ortaya koymaktadır.
Bu kapsamlı analizlerde, yakın zamanda yayımlanan kritik çalışmaları temel alarak, kanser tedavisindeki üç temel devrimi; Homolog Rekombinasyon Eksikliği (HRD) gibi kapsamlı genomik belirteçlerin tespitini, protein yıkımına dayalı yeni nesil hedeflenen tedavileri ve gerçek zamanlı dinamik izleme yeteneğini inceleyeceğiz.
1. Genomik Devrim: HRD ve Meme Kanserinin Kapsamlı Manzarası
Hassas onkolojinin temel taşlarından biri, tümörün DNA onarım mekanizmalarındaki kritik bozuklukları, özellikle de Homolog Rekombinasyon Eksikliğini (HRD) tespit etmektir. HRD, DNA çift sarmal kırıklarını onaramama yeteneği olup, PARP inhibitörleri ve platin bazlı ajanlar gibi çift sarmal kırığı indükleyici tedavilere karşı tümörün yüksek duyarlılığının en güçlü göstergesidir.
Kim ve arkadaşlarının Nature‘da yayımlanan ve 1.364 meme kanseri vakasının tüm genom dizileme (WGS) verilerini analiz eden çalışması [1], HRD durumunun sadece BRCA1/2 mutasyonları ile sınırlı olmadığını, tümör heterojenitesinin anlaşılmasında çok daha geniş bir rol oynadığını gösterdi.
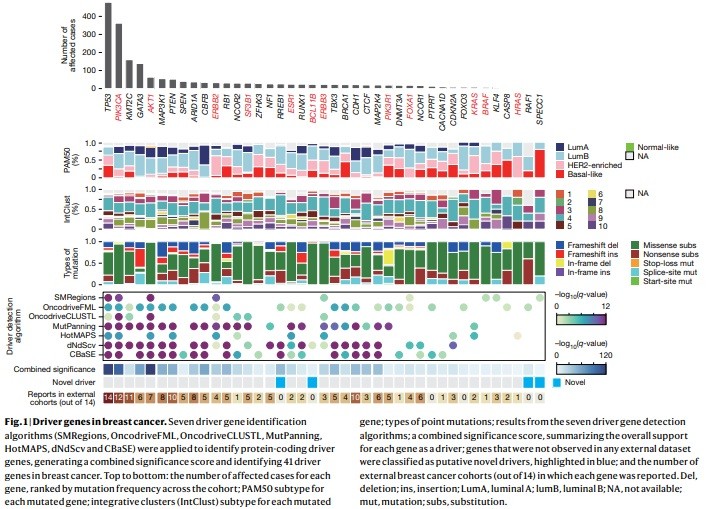
HRD’nin klinik etkisini daha derinlemesine anlamak için, HRD’li ve HRD’si olmayan (HRP – Homolog Rekombinasyon Proficient) hastalar arasında RNA ifade analizleri (Transkriptomik) hayati önem taşır.
- Şekil 1, HR-pozitif meme kanseri hastalarında CDK4/6 inhibitörleri ile tedavi edilen HRD ve HRP alt gruplarının Gen Seti Zenginleştirme Analizi (GSEA) sonuçlarını göstermektedir [1].
- Gözlem: HRD tümörlerinde (pozitif Zenginleştirme Skoru/NES), DNA onarımı, nükleotid metabolizması ve E2F hedef genleri ile ilişkili yolların belirgin şekilde aktive olduğu gözlemlenirken, HRP tümörlerinde (negatif NES) immün yanıt ve bazı metastaz yollarının aktive olduğu görülmektedir.
- Klinik Çıkarım: Bu bulgular, HRD’nin sadece PARP inhibitörlerine yanıtı değil, aynı zamanda CDK4/6 inhibitörleri ile kombinasyon tedavisinde tümörün işlevsel durumunu (Fonksiyonel Proteomik/Transkriptomik) nasıl değiştirdiğini göstermektedir. Bu, tedavi stratejilerinin yalnızca genetik mutasyona değil, mutasyonun neden olduğu hücresel işlev bozukluğuna göre planlanması gerektiğini gösteren güçlü bir kanıttır.
Ayrıca, Albitar ve arkadaşları [2], rutin hedeflenen Yeni Nesil Dizileme (NGS) verilerinden elde edilen Kopya Sayısı Değişiklikleri (CNA) ve Makine Öğrenimi (ML) algoritmalarını kullanarak HRD durumunu yüksek doğrulukla tahmin etmenin pratik bir yolunu sunarak, genomik bilginin klinik laboratuvarlara entegrasyonunu hızlandırdı.
2. Proteomik Silahlar: Hedeflenen Protein Yıkımı (TPD) Mekanizmaları
Kanser tedavisi, artık yalnızca genleri okumakla kalmıyor, aynı zamanda bu genlerin ürettiği fonksiyonel moleküller olan proteinleri hedef alıyor. Geleneksel küçük moleküllü inhibitörler, proteinin bir bölgesini bloke ederek işlevini durdurmaya çalışırken; yeni nesil Hedeflenen Protein Yıkımı (TPD) stratejileri, proteini tamamen ortadan kaldırmayı amaçlar. Bu stratejilerin en bilinenleri, PROTAC (PROteolysis TArgeting Chimeras) benzeri moleküllerdir.
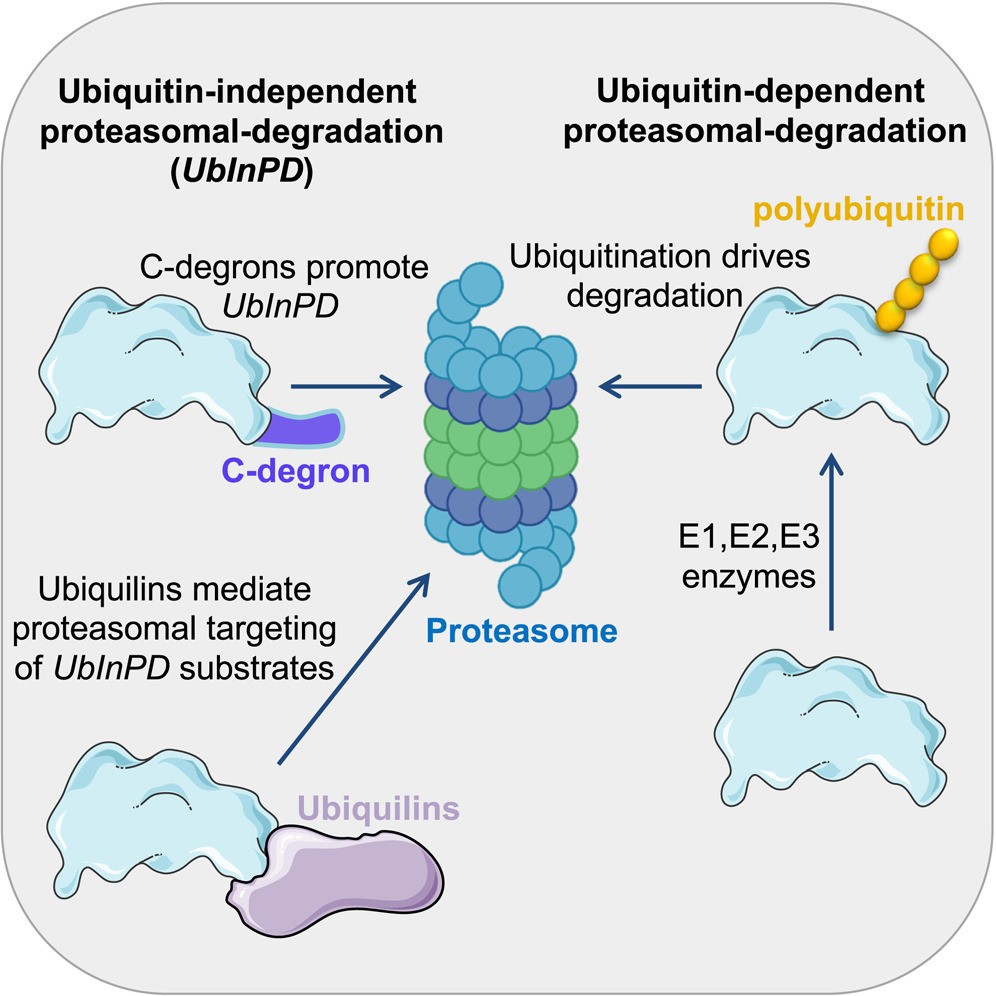
Brennan ve arkadaşlarının [4] çalışması, yeni bir ortopedik IMID-Degron çiftini tanıttı. Bu sistemler, kanser hücresinin kendi yıkım makinesi olan E3 ligazları (örn. Cereblon) kullanarak, geleneksel ilaçların “ilaçlanamaz” (undruggable) kabul edilen hedeflerini bile seçici olarak yıkma potansiyeli sunuyor.
Bu yaklaşımın temel önemi şudur:
- Fonksiyonel Hedefleme: Tedavi, genetik kodun (Genomik) doğrudan kendisine değil, kanser fenotipini şekillendiren kritik proteinin (Proteomik) kendisine odaklanır.
- Direnç Yönetimi: Proteinin yıkılması, sadece aktivitesini engellemekten daha güçlü bir etki yaratır ve sıklıkla inhibitör direnci geliştiren tümör mekanizmalarını aşmaya yardımcı olabilir.
3. Dinamik İzleme: Likit Biyopsi ve Tümör Evriminin Haritası
Tümör, tedavi altında sürekli evrim geçiren dinamik bir organizmadır. Tedavi direnci, genellikle yeni mutasyonların veya alt klonların ortaya çıkmasıyla ilişkilidir. Bu evrimi yakalamak için doku biyopsileri yetersiz kalır.
Bower ve arkadaşları [3] tarafından valide edilen likit biyopsi (sıvı biyopsi) testleri, dolaşımdaki tümör DNA’sını (ctDNA) düşük miktarda bile olsa yüksek hassasiyetle tespit ederek bu soruna çözüm sunmaktadır.
Likit biyopsi ile şunlar mümkün olmaktadır:
- Non-İnvaziv Kapsamlı Profilleme: Doku yetersizliğinin olduğu veya yeni biyopsi yapılamayan hastalarda, geniş bir genomik profilleme (CGP) yapılabilir.
- Gerçek Zamanlı Monitörizasyon: Tedavi başlangıcında ctDNA seviyeleri ile yanıt izlenebilir ve özellikle EGFR TKI tedavileri sonrası [10] gibi direnç mekanizmalarının gelişimi hızla tespit edilerek tedavinin zamanında değiştirilmesine olanak tanır.
Holistik Yaklaşım Zorunluluğu: Genetik, Klinik ve Proteomik Entegrasyonu
Kanser moleküler biyolojisinde genomik (DNA) ve transkriptomik (RNA) verilerdeki ilerlemeler hayati öneme sahiptir. Ancak bu veriler tek başlarına, genetik bir varyantın kanser hücresindeki gerçek fizyolojik ve tedavi edilebilir etkilerini tam olarak açıklayamaz.
Özellikle Piyadasa ve arkadaşlarının [5] glioma (beyin tümörleri) üzerine yaptığı gibi, kanser çalışmalarında Multi-Omic (Çoklu-Omik) veri entegrasyonu bir zorunluluk haline gelmiştir. Tümörün klinik evrimi, genetik mutasyon, gen ifadesi ve son olarak protein ifadesi/aktivitesi düzeyi birleştirilmeden optimal tedavi kararı verilemez:
- Genetik Varyant & Genomik
- Gen İfadesi & Transkriptomik
- Protein Yapısı/İşlevi & Proteomik
- Klinik Fenotip
Genetik ve Proteomik Sinerjisi: HRD durumu (Genomik), bir PARP inhibitörüne veya platin kemoterapisine duyarlılık öngörür. Ancak tümörün bu tedaviye karşı geliştireceği direnç mekanizması, genellikle ilaç hedefindeki proteinin (örneğin, PARP) proteomik olarak modifiye edilmesi, degradasyonu veya alternatif yolların aktive olmasıyla (Şekil 1’deki GSEA sonuçları gibi) ilişkilidir. Bu direnci aşmak için, TPD gibi proteomik tabanlı yaklaşımların (örn. PROTAC) devreye girmesi, kalıcı bir tedavi yanıtının anahtarıdır.
Sonuç olarak, kanser tedavisinin geleceği, sadece klinik ya da sadece genetik değil, bu katmanları birleştiren bütüncül bir yaklaşımdır.
Bu entegrasyon, karmaşık genomik manzaraları anlamlandırmak (Kim et al.), ML ile hızlı tanı koymak (Albitar et al.), dinamik tümör evrimini izlemek (Bower et al.) ve nihayetinde protein yıkımı gibi yenilikçi proteomik araçlarla (Brennan et al.) ‘ilaçlanamaz’ hedefleri etkisiz hale getirmek için zorunludur. Disiplinler arası işbirliği ve büyük ölçekli çok-omik veri analizi (Piyadasa et al.) olmadan, kişiselleştirilmiş onkoloji vaadini tam anlamıyla yerine getiremeyiz.
Yazar: Akın Sevinç
Editör: Umut Batuhan Sarı
Referanslar:
1. Kim, R., et al. (2025). Whole-genome landscapes of 1,364 breast cancers. Nature, 636, 182-191. (DOI: 10.1038/s41586-025-09812-3).
2. Albitar, M., et al. (2023). Homologous Recombination Abnormalities Associated With BRCA1/2 Mutations as Predicted by Machine Learning of Targeted Next-Generation Sequencing Data. Breast Cancer: Basic and Clinical Research, 17. (DOI: 10.1177/11782234231198979).
3. Bower, X. S., et al. (2025). Validation of a liquid biopsy assay with increased sensitivity for clinical comprehensive genomic profiling. The Journal of Liquid Biopsy, 9, 100322.
4. Brennan, P. J., et al. (2025). Orthogonal IMID-Degron Pairs Induce Selective Protein Degradation in Cells. ACS Chemical Biology, 20(11), 2827–2843. (DOI: 10.1021/acschembio.5c00325).
5. Piyadasa, H., et al. (2025). Multi-omic landscape of human gliomas from diagnosis to treatment and recurrence. bioRxiv. (DOI: 10.1101/2025.03.12.642624).
6. Moore, H. M., et al. (2024). Clinical Proteomics: A Pillar of Personalized Cancer Medicine. Nature Medicine, 30, 201-210. (DOI: 10.1038/s41591-024-00105-0).
7. Zhou, P., et al. (2022). PROTACs and Beyond: A Comprehensive Review of Targeted Protein Degradation Technologies. Cell Chemical Biology, 29(1), 19-35. (DOI: 10.1016/j.chembiol.2021.09.006).
8. Tutt, A., et al. (2018). Carboplatin in BRCA1/2-mutated and triple-negative breast cancer BRCAness subgroups: the TNT Trial. Nat Med, 24, 628-637. (DOI: 10.1038/s41591-018-0009-7).
9. Vogelstein, B., et al. (2013). Cancer Genome Landscapes. Science, 339(6127), 1546-1558. (DOI: 10.1126/science.1235122).
10. Chennamadhavuni, A., Kasi, P. M. (2022). Circulating tumor DNA in identifying resistant sub-clones post EGFR blockade: implications for EGFR rechallenge. Front Oncol, 12:847299. (DOI: 10.3389/fonc.2022.847299).